Description
Nous détectons et quantifions les acides gras phospholipidiques (PLFA) qui sont utilisés pour le profilage écologique des échantillons de sol.
Nous réalisons :
- Extraction et dérivatisation des PLFA du sol,
- Analyse GC-MS/FID,
- Rapport sur les profils des communautés microbiennes du sol (Rapport d'écologie microbienne du sol). Cliquez ici pour consulter un exemple de rapport sur l'“ Écologie microbienne des sols ”..
Notre liste d'acides gras détectables et quantifiables comprend :
- acides gras insaturés (mono et poly)
- Acides gras ramifiés (iso, antéiso et à chaîne moyenne)
- Acides gras cycliques.
- Acides gras saturés (C2-C36)
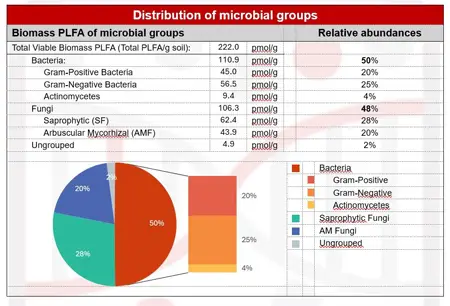
Les groupes de communautés microbiennes pour lesquels nous disposons de biomarqueurs PLFA comprennent (la liste des biomarqueurs PLFA mentionnés s'allonge constamment) :
- Bactéries : 15 biomarqueurs PLFA, répartis entre les sous-groupes bactériens
- Gram positif : 7 biomarqueurs PLFA.
- Gram négatif : 4 biomarqueurs PLFA.
- Actinomycètes : 3 biomarqueurs PLFA.
- Bactéries oxydant les nitrites (NOB) : 1 biomarqueur PLFA.
- Champignons : 3 biomarqueurs
- Champignons saprophytes : 2 biomarqueurs
- Champignons mycorhiziens arbusculaires (CMA) : 1 biomarqueur
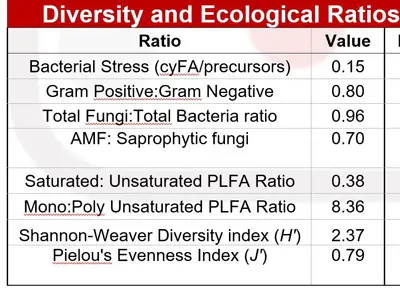
Nous calculons également les indices de diversité, les ratios écologiques et les indicateurs physiologiques (de stress) suivants, basés sur les PLFA :
- Indicateur de stress bactérien (rapport acides gras cycliques/leurs précurseurs)
- Rapport Gram positif/Gram négatif
- Rapport champignons/bactéries
- Rapport champignons arbusculaires/champignons saprophytes
- Rapport PLFA saturés/insaturés.
- Rapport PLFA mono-insaturé/polyinsaturé
- Indice de diversité de Shannon-Weaver (H'’)
- L'indice d'équitabilité de Pielou (J'’)